#lectura de datos
tabla.hatco <- suppressMessages(read_csv("data/tabla.hatco.anacor.csv"))
#eliminamos la primera columna (específico de paquete `ca` con el que trabajaremos que necesita `rownames`)
tabla.hatco.ca <- subset(tabla.hatco, select = -1)
#asignamos los nombres de fila con la propiedad rownames, pues así lo necesita el paquete `ca`.
rownames(tabla.hatco.ca) = c("x1", "x2", "x3", "x4", "x5", "x6", "x7", "x8")5 Correspondencias simple - S03
5.1 Introducción a la técnica
El análisis de correspondencias es una técnica de interdependencia cuyo objetivo es la representación de las relaciones bidimensionales derivadas de la relación entre dos variables o de un conjunto matricial de datos ordenados. Trabaja con variables no métricas aunque el contenido medido sea métrico (frecuencias, sumas, medias), pues realmente nos importan sus categorías o niveles, siendo posible utilizar la recodificación para obtener niveles de medida con los que trabajar. Se muestra para visualizar los diferentes niveles de medidas no métricas por lo que a menudo se le conoce como el análisis de representación de tablas de contingencia trabajando con el concepto de frecuencias relativas y distancias.
5.1.1 Transformación de datos cuantitativos a cualitativos
En muchos casos se hace necesario obtener la tabla de frecuencias de partida utilizando algunas estrategias de cálculo, disponiendo de esta forma adecuada los datos:
- Cálculos de dicotomías
- Clasificación en intervalos
- Establecimiento de condiciones de corte
Nuestro objetivo será la obtención de una tabla de doble entrada; en ocasiones la fuente de datos es ya elaborada de forma ajena a nuestro trabajo, es una tabla de un periódico o libro y se debe introducir al software usado. Para hacer esto, podemos crear un fichero con tres variables: variable fila, variable columna y peso en SPSS (ver siguiente) o la lectura de de la tabla o matriz de datos en R desde fichero texto, utilizando por ejemplo el paquete readr.
Este es el método por el que optamos.
El análisis de correspondencias no es exigente con las propiedades estadísticas de los datos. Como ya indicamos, utiliza variables no métricas en su forma más simple (tabla de contingencia), pero sin embargo hay que ser cuidadoso respecto al sentido del análisis en función de la representación de los datos.
En nuestro ejemplo pensemos que …
- Los poseedores de los atributos son comparables respecto a esos atributos.
- Confirmar que las empresas A,B, …, I son efectivamente competidoras de HATCO.
- Que basan su diferenciación en los atributos X1 a X8.
- El listado de atributos debe ser exhaustivo y no dejamos ninguno relevante para la caracterización de las empresas.
Comenzamos por ver la relevancia de la tabla, validar si realmente hay un cierto nivel de dependencia entre las categorías de fila y las categorías de columna. Para ello utilizamos la prueba Chi2.
5.2 Prueba Chi2 de homogeneidad
Para disponer de un indicador del ajuste de dependencia u homogeneidad, utilizamos la prueba de Chi2 que la vimos en anteriores análisis. Cuánto mayor es el valor de Chi2 mayores son las discrepancias entre observado y esperado. Sin embargo, sólo somos capaces de dimensionar en valor relativo cuando vemos otros indicadores como Phi o V de Cramer, que también serían posibles si el objeto fuera tabla calculada, pero en nuestro caso es leída directa, por lo que aplicamos las transformaciones de fichero a matrix y de matriz a table. No calculamos Phi porque no es una tabla de 2*2.
tabla.hatco.ca <- as.table(as.matrix(tabla.hatco.ca))
crosstable_statistics(tabla.hatco.ca)
# Measure of Association for Contingency Tables
Chi-squared: 122.6006
Cramer's V: 0.1435
df: 63
p-value: < .001***
Observations: 8515.3 Nuestro ejemplo
HATCO quiere identificar a sus principales competidores:
- Quiere hacerlo en función de su posición respecto a las principales variables de competencia:
- X1 Rapidez del servicio
- X2 Nivel de precios
- X3 Flexibilidad de precios
- X4 Imagen del fabricante
- X5 Calidad del servicio
- X6 Imagen de los vendedores
- X7 Calidad del producto
- X8 Tamaño de la empresa
- La medida que se utiliza es la asociación de la característica con el competidor. Para ello se ha utilizado una transformación de la medición original a una caracterización (0/1).
- En nuestro ejemplo: en la tabla de contingencia, una frecuencia de 1 implica que ese competidor se ha señalado como “que dispone de la característica competitiva” en la característica o atributo de fila.
5.4 Análisis de correspondencias simple (Michael Greenacre)
Calculamos utilizando el paquete ca de Michael Greenacre. Sus resultados son semejantes a SPSS pero el mapa sale invertido en cuadrantes, lo que no implica distinta interpretación.
5.4.1 Resumen de resultados
Obtenemos los resultados más característicos con la función ca(). Almacenamos el objeto para ir mostrando después más completos sus resultados
res <- ca(tabla.hatco.ca)
res
Principal inertias (eigenvalues):
1 2 3 4 5 6 7
Value 0.076541 0.047813 0.015291 0.002658 0.000806 0.000576 0.000381
Percentage 53.13% 33.19% 10.61% 1.84% 0.56% 0.4% 0.26%
Rows:
x1 x2 x3 x4 x5 x6 x7
Mass 0.146886 0.144536 0.098707 0.118684 0.123384 0.124559 0.081081
ChiDist 0.199723 0.088497 0.580707 0.400294 0.285309 0.386846 0.808039
Inertia 0.005859 0.001132 0.033286 0.019017 0.010044 0.018640 0.052940
Dim. 1 0.388333 0.219066 0.083693 -1.285544 -0.383784 -0.836930 2.863462
Dim. 2 0.524538 -0.097621 -2.641197 0.610370 1.073949 0.211061 0.636902
x8
Mass 0.162162
ChiDist 0.139329
Inertia 0.003148
Dim. 1 -0.153942
Dim. 2 -0.524856
Columns:
hatco a b c d e f
Mass 0.108108 0.119859 0.089307 0.079906 0.078731 0.123384 0.113984
ChiDist 0.287008 0.321688 0.430028 0.569393 0.402272 0.184239 0.257577
Inertia 0.008905 0.012403 0.016515 0.025906 0.012740 0.004188 0.007562
Dim. 1 -0.470333 -1.021735 -0.843200 1.933384 0.969935 -0.449822 -0.838114
Dim. 2 0.626124 0.580139 -1.583295 -0.793574 -1.189113 0.502451 0.446827
g h i
Mass 0.103408 0.076381 0.106933
ChiDist 0.532696 0.491456 0.274442
Inertia 0.029344 0.018448 0.008054
Dim. 1 1.680687 -0.392286 0.233423
Dim. 2 1.092070 -1.944341 0.784252Para poder representar gráficamente los datos de la tabla, se debe reducir la dimensionalidad, puesto que solo vamos a ser capaces de ver los datos en mapas de dos dimensiones, o como mucho en tres. El mapa explica en sus dos primeras dimensiones el 86,3% de la información original (suma de las dos primeras dimensiones).
Podemos luego combinar para otras dimensiones, 1-3, 2-5 pero es muy poco habitual, dado que la pérdida de información (y por tanto la dificultad de explicación) es muy alta.
Analicemos ahora poco a poco los elementos que componen el análisis.
Algunos datos relevantes son:
- Llamamos perfil al vector formado por los elementos de columna y/o fila.
- Llamamos perfil medio al perfil total de columna y/o al perfil total de columna.
Otra forma de verlo:
* Las coordenadas en el mapa se calculan utilizando algunos conceptos típicos en el análisis multivariante:
* La inercia total, o medida de dispersión entre los perfiles y el perfil promedio, es un indicador de la dispersión o falta de correspondencia entre un punto fila y/o columna. Se calcula como el valor de Chi-cuadrado cociente con el total de casos.- La hipótesis nula es una transformación de la estándar, el perfil medio sería la homogeneidad de los perfiles (Ho en Chi-cuadrado es la independencia).
- Homogeneidad = Independencia.
- Heterogeneidad = Dependencia.
5.4.1.0.1 Valor singular
res[["sv"]] #extraemos el elemento llamado sv[1] 0.27666126 0.21866273 0.12365880 0.05155155 0.02838433 0.02400166 0.019514415.4.1.1 Análisis de las filas
5.4.1.1.1 Nombre de las filas (atributos)
res[["rownames"]][1] "x1" "x2" "x3" "x4" "x5" "x6" "x7" "x8"5.4.1.1.2 Masa de las filas (perfil)
Perfiles de las filas, son los porcentajes horizontales, aunque no se llaman así porque en muchos casos las columnas y/o filas de la tabla no son realmente categorías de una misma variable (frecuencias relativas). Llamados masa, es el cálculo de los porcentajes marginales (sin contar con la otra variable), denominado en algunos casos el perfil medio o también centroide o baricentro. Es decir el peso del atributo o de la empresa en el total marginal.
res[["rowmass"]][1] 0.14688602 0.14453584 0.09870740 0.11868390 0.12338425 0.12455934 0.08108108
[8] 0.162162165.4.1.1.3 Distancia de las filas
La representación de los datos de la tabla en el mapa se hace atendiendo al cálculo de distancias. El concepto de distancia, en este caso la distancia Chi-cuadrado o cálculo de la distancia euclídea entre los vectores fila y su masa de fila.
res[["rowdist"]][1] 0.19972301 0.08849696 0.58070663 0.40029405 0.28530854 0.38684579 0.80803871
[8] 0.139329075.4.1.1.4 Inercia de las filas
La inercia de cada perfil se calcula como producto de la masa por el cuadrado de la distancia chi-cuadrado de ese perfil al promedio. La inercia mide lo lejos que se hallan los perfiles fila o columna de su perfil medio.
res[["rowinertia"]][1] 0.005859178 0.001131963 0.033286129 0.019017354 0.010043597 0.018640264
[7] 0.052939991 0.0031479885.4.1.1.5 Coordenadas de las filas
Punto de ubicación en el mapa de cada punto fila.
res[["rowcoord"]] Dim1 Dim2 Dim3 Dim4 Dim5 Dim6
x1 0.38833337 0.52453784 -0.48064888 2.0327480 -0.7993536 -0.3021486
x2 0.21906639 -0.09762105 -0.05138317 -0.4795627 -1.3748826 0.7323587
x3 0.08369285 -2.64119673 -0.34019450 0.2493870 0.6347806 -1.2085960
x4 -1.28554439 0.61037038 -0.89764463 0.4026826 1.7762794 0.8684848
x5 -0.38378371 1.07394894 -0.73674199 -1.3240795 -0.2697071 -1.8311119
x6 -0.83693026 0.21106121 2.47591880 0.1896399 0.1476687 -0.2958822
x7 2.86346220 0.63690241 0.48320469 -0.4465221 1.4975360 0.2144778
x8 -0.15394237 -0.52485592 -0.23761538 -0.7752912 -0.3939105 1.2342381
Dim7
x1 0.53731697
x2 -1.78938738
x3 -0.32626826
x4 -0.72333444
x5 0.28708482
x6 0.08839279
x7 -0.08239776
x8 1.591052495.4.1.2 Análisis de las columnas
5.4.1.2.1 Nombre de las columnas (las marcas)
res[["colnames"]] [1] "hatco" "a" "b" "c" "d" "e" "f" "g" "h"
[10] "i" 5.4.1.2.2 Masa de las columnas (perfil)
Perfiles de las columnas, son los porcentajes verticales, aunque no se llaman así porque en muchos casos las columnas y/o filas de la tabla no son realmente categorías de una misma variable (frecuencias relativas). Llamados masa, es el cálculo de los porcentajes marginales (sin contar con la otra variable), denominado en algunos casos el perfil medio o también centroide o baricentro. Es decir el peso del atributo o de la empresa en el total marginal.
res[["colmass"]] [1] 0.10810811 0.11985899 0.08930670 0.07990599 0.07873090 0.12338425
[7] 0.11398355 0.10340776 0.07638073 0.106933025.4.1.2.3 Distancia de las columnas
La representación de los datos de la tabla en el mapa se hace atendiendo al cálculo de distancias. El concepto de distancia, en este caso la distancia Chi-cuadrado o cálculo de la distancia euclídea entre los vectores columna y su masa de columna.
res[["coldist"]] [1] 0.2870082 0.3216884 0.4300280 0.5693934 0.4022721 0.1842392 0.2575766
[8] 0.5326961 0.4914556 0.27444165.4.1.2.4 Inercia de las columnas
La inercia de cada perfil se calcula como producto de la masa por el cuadrado de la distancia chi-cuadrado de ese perfil al promedio. La inercia mide lo lejos que se hallan los perfiles fila o columna de su perfil medio.
res[["colinertia"]] [1] 0.008905264 0.012403421 0.016514959 0.025906232 0.012740460 0.004188165
[7] 0.007562317 0.029343514 0.018448129 0.0080540005.4.1.2.5 Coordenadas de las columnas
Punto de ubicación en el mapa de cada punto columna.
res[["colcoord"]] Dim1 Dim2 Dim3 Dim4 Dim5 Dim6
hatco -0.4703327 0.6261240 -1.6935356 0.29465670 -1.7292066 0.4655331
a -1.0217346 0.5801388 0.5338336 -0.07823246 1.1100906 1.6894406
b -0.8432000 -1.5832948 0.7964316 0.26209544 0.2874627 -0.1659282
c 1.9333842 -0.7935744 -0.1648613 1.59150233 -0.5015929 1.0023065
d 0.9699347 -1.1891134 1.1762320 0.24157388 -0.3215290 0.1038722
e -0.4498224 0.5024514 0.2356379 1.01941582 0.2256877 -2.0473172
f -0.8381141 0.4468266 -0.2145714 0.65967745 0.3110986 0.1309981
g 1.6806871 1.0920696 -0.6758338 -0.77145803 1.5096436 -0.3224750
h -0.3922857 -1.9443407 -1.6117961 -1.83337906 0.3985224 -0.4838085
i 0.2334232 0.7842522 1.4674729 -1.62005491 -1.4611537 -0.1711086
Dim7
hatco -0.4460250
a -1.1173385
b 1.3363279
c 0.3955033
d -1.4550986
e -0.9157056
f 1.6885279
g 0.4368324
h -0.4346288
i 0.50781095.4.2 Representación gráfica: los mapas de puntos
plot(res)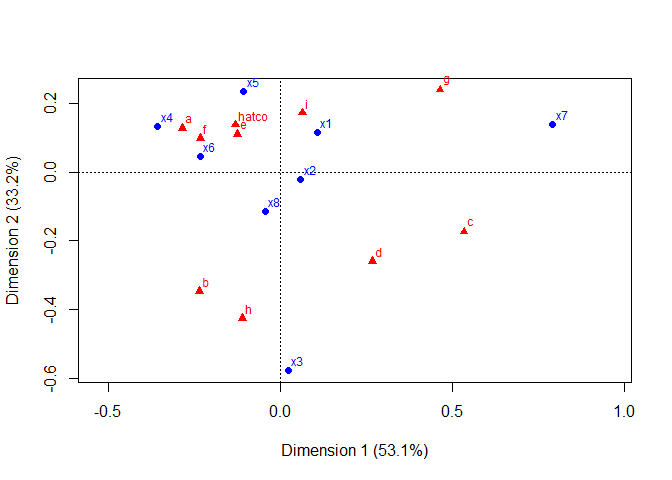
plot(res, lines=TRUE)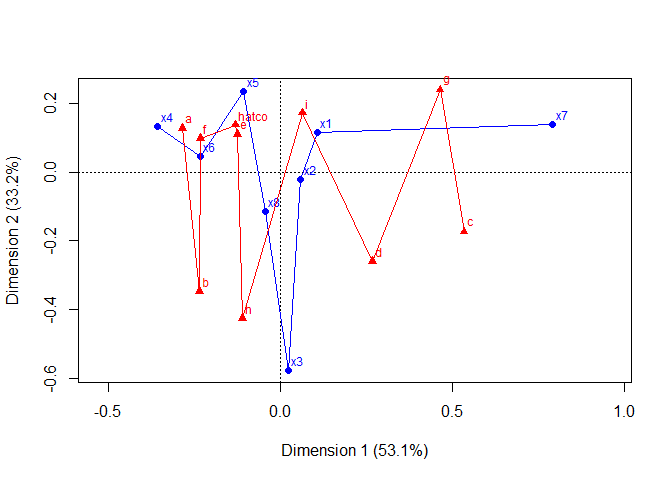
plot(res, arrows=c(TRUE, TRUE))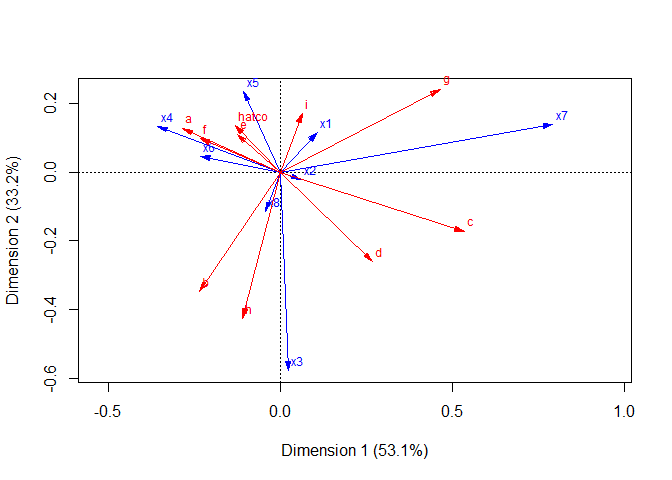
5.5 Análisis de correspondencias (CA de FactoMineR)
Ahora hacemos el cálculo con el algoritmo CA (nótese diferencia por mayúsculas) del paquete FactoMineR. Generamos una salida de tabla en modo gráfico, donde visualmente el diámetro de la circunferencia es mayor cuanto mayor sea el valor de la celda
dtmatrix <- as.matrix(tabla.hatco.ca)
dttable <- as.table(dtmatrix)
balloonplot(t(dttable),main = "Empresa * Atributo",xlab = "Empresa",ylab = "Atributo",label = TRUE,show.margins = TRUE)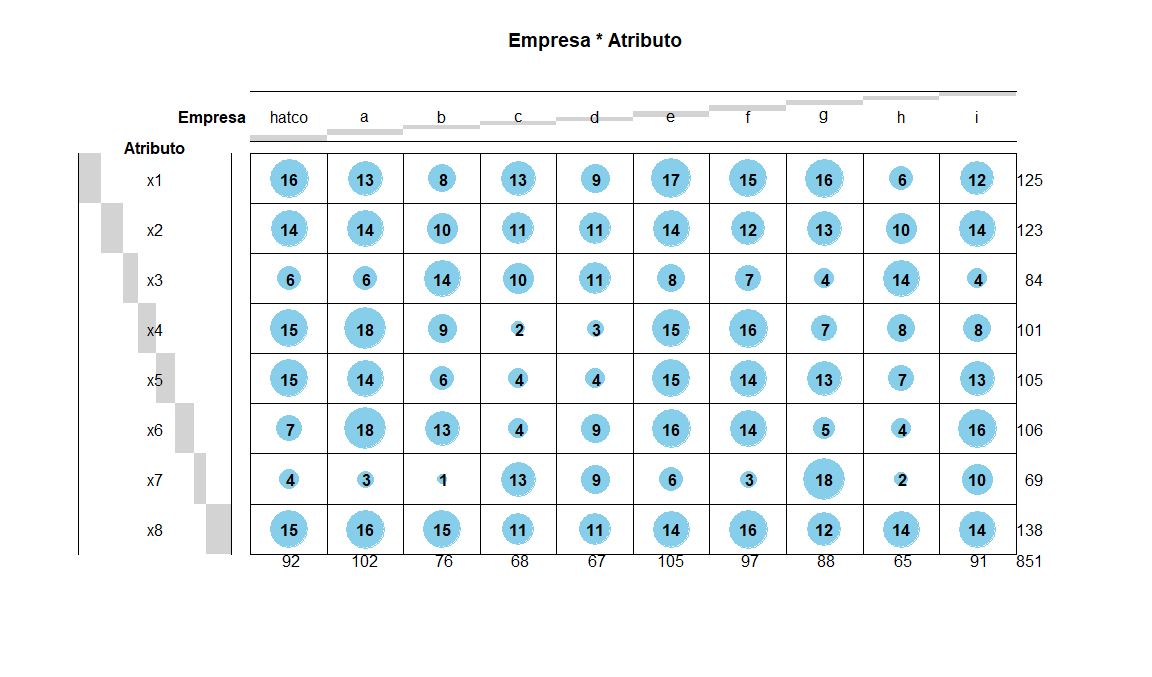
5.5.1 Resultados
Calculamos el análisis de correspondencias del paquete FactoMineR. Se usa la tabla original.
Gráfico básico
res.ca <- CA(tabla.hatco.ca, graph = TRUE)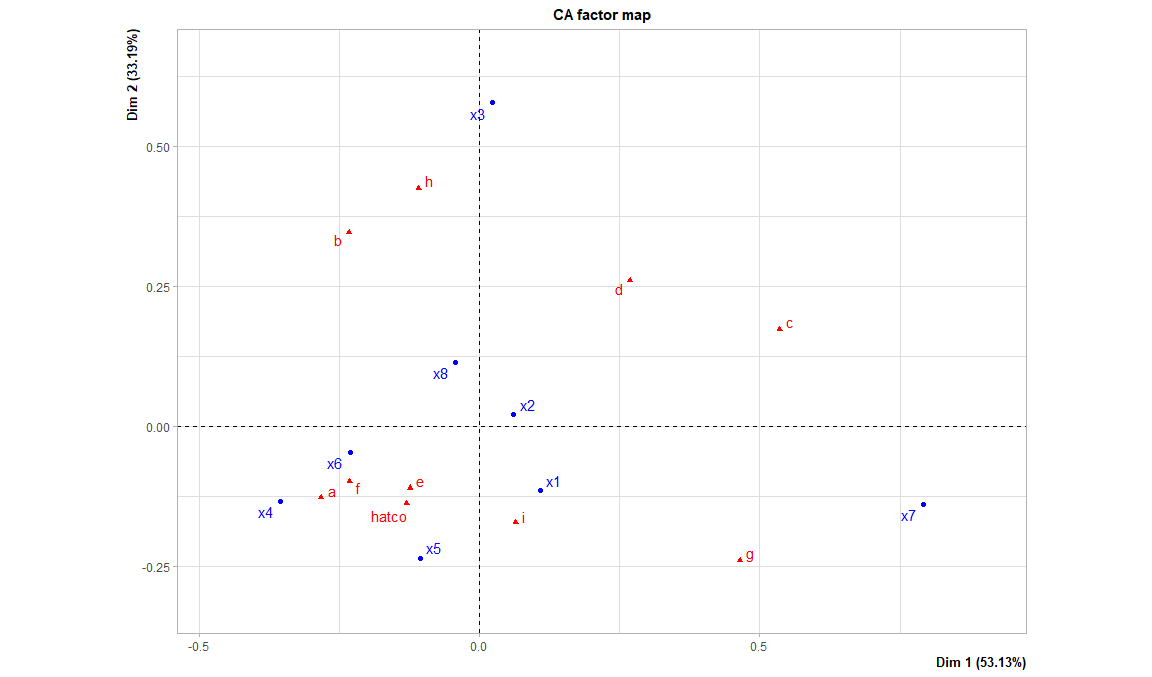
Valores propios
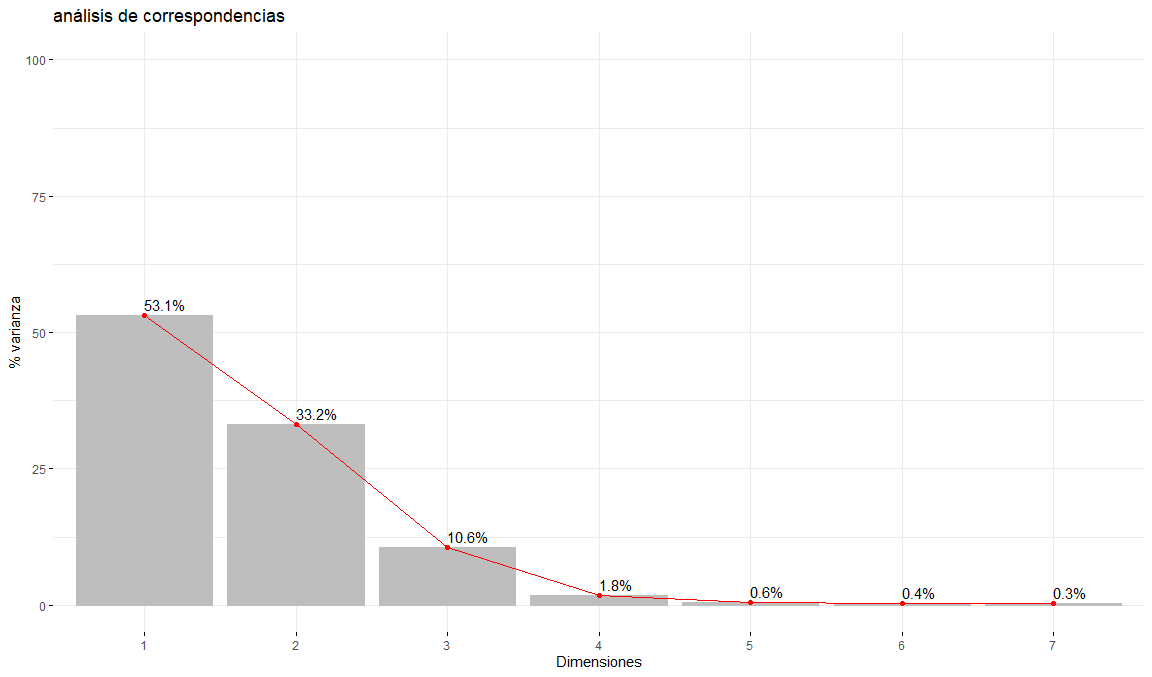
res.ca[["eig"]] eigenvalue percentage of variance cumulative percentage of variance
dim 1 0.0765414510 53.1292637 53.12926
dim 2 0.0478133897 33.1884248 86.31769
dim 3 0.0152914984 10.6141972 96.93189
dim 4 0.0026575621 1.8446778 98.77656
dim 5 0.0008056703 0.5592352 99.33580
dim 6 0.0005760795 0.3998706 99.73567
dim 7 0.0003808120 0.2643308 100.00000Masa de las columnas
round(res.ca[["call"]][["marge.col"]], 3)hatco a b c d e f g h i
0.108 0.120 0.089 0.080 0.079 0.123 0.114 0.103 0.076 0.107 Masa de las filas
round(res.ca[["call"]][["marge.row"]], 3) x1 x2 x3 x4 x5 x6 x7 x8
0.147 0.145 0.099 0.119 0.123 0.125 0.081 0.162 Coordenadas de las filas
round(res.ca[["row"]][["coord"]], 3) Dim 1 Dim 2 Dim 3 Dim 4 Dim 5
x1 0.107 -0.115 0.059 -0.105 0.023
x2 0.061 0.021 0.006 0.025 0.039
x3 0.023 0.578 0.042 -0.013 -0.018
x4 -0.356 -0.133 0.111 -0.021 -0.050
x5 -0.106 -0.235 0.091 0.068 0.008
x6 -0.232 -0.046 -0.306 -0.010 -0.004
x7 0.792 -0.139 -0.060 0.023 -0.043
x8 -0.043 0.115 0.029 0.040 0.011Contribuciones de las filas
round(res.ca[["row"]][["contrib"]], 3) Dim 1 Dim 2 Dim 3 Dim 4 Dim 5
x1 2.215 4.041 3.393 60.694 9.386
x2 0.694 0.138 0.038 3.324 27.322
x3 0.069 68.857 1.142 0.614 3.977
x4 19.614 4.422 9.563 1.924 37.447
x5 1.817 14.231 6.697 21.632 0.898
x6 8.725 0.555 76.357 0.448 0.272
x7 66.482 3.289 1.893 1.617 18.183
x8 0.384 4.467 0.916 9.747 2.516Calidad de las filas
round(res.ca[["row"]][["cos2"]], 3) Dim 1 Dim 2 Dim 3 Dim 4 Dim 5
x1 0.289 0.330 0.089 0.275 0.013
x2 0.469 0.058 0.005 0.078 0.194
x3 0.002 0.989 0.005 0.000 0.001
x4 0.789 0.111 0.077 0.003 0.016
x5 0.138 0.677 0.102 0.057 0.001
x6 0.358 0.014 0.626 0.001 0.000
x7 0.961 0.030 0.005 0.001 0.003
x8 0.093 0.678 0.044 0.082 0.006Inercia de las filas
round(res.ca[["row"]][["inertia"]], 3)[1] 0.006 0.001 0.033 0.019 0.010 0.019 0.053 0.003Coordenadas de las columnas
round(res.ca[["col"]][["coord"]], 3) Dim 1 Dim 2 Dim 3 Dim 4 Dim 5
hatco -0.130 -0.137 0.209 -0.015 0.049
a -0.283 -0.127 -0.066 0.004 -0.032
b -0.233 0.346 -0.098 -0.014 -0.008
c 0.535 0.174 0.020 -0.082 0.014
d 0.268 0.260 -0.145 -0.012 0.009
e -0.124 -0.110 -0.029 -0.053 -0.006
f -0.232 -0.098 0.027 -0.034 -0.009
g 0.465 -0.239 0.084 0.040 -0.043
h -0.109 0.425 0.199 0.095 -0.011
i 0.065 -0.171 -0.181 0.084 0.041Contribuciones de las columnas
round(res.ca[["col"]][["contrib"]], 3) Dim 1 Dim 2 Dim 3 Dim 4 Dim 5
hatco 2.391 4.238 31.006 0.939 32.326
a 12.513 4.034 3.416 0.073 14.770
b 6.350 22.388 5.665 0.613 0.738
c 29.869 5.032 0.217 20.239 2.010
d 7.407 11.132 10.893 0.459 0.814
e 2.497 3.115 0.685 12.822 0.628
f 8.007 2.276 0.525 4.960 1.103
g 29.210 12.333 4.723 6.154 23.567
h 1.175 28.875 19.843 25.674 1.213
i 0.583 6.577 23.028 28.065 22.830Calidad de las columnas
round(res.ca[["col"]][["cos2"]], 3) Dim 1 Dim 2 Dim 3 Dim 4 Dim 5
hatco 0.206 0.228 0.532 0.003 0.029
a 0.772 0.156 0.042 0.000 0.010
b 0.294 0.648 0.052 0.001 0.000
c 0.882 0.093 0.001 0.021 0.001
d 0.445 0.418 0.131 0.001 0.001
e 0.456 0.356 0.025 0.081 0.001
f 0.810 0.144 0.011 0.017 0.001
g 0.762 0.201 0.025 0.006 0.006
h 0.049 0.748 0.164 0.037 0.001
i 0.055 0.390 0.437 0.093 0.023Inercia de las columnas
round(res.ca[["col"]][["inertia"]], 3) [1] 0.009 0.012 0.017 0.026 0.013 0.004 0.008 0.029 0.018 0.0085.5.2 Calidad
Calculamos el gráfico de las calidad de la varianza explicada por las dimensiones, descendente por representación.
fviz_eig(
res.ca,
choice = c("variance", "eigenvalue"),
geom = c("bar", "line"),
barfill = "grey",
barcolor = "grey",
linecolor = "red",
addlabels = TRUE,
hjust = 0,
main = "análisis de correspondencias",
xlab = "Dimensiones",
ylab = "% varianza",
ggtheme = theme_minimal(),
ylim = c(0, 100),
repel = FALSE
)
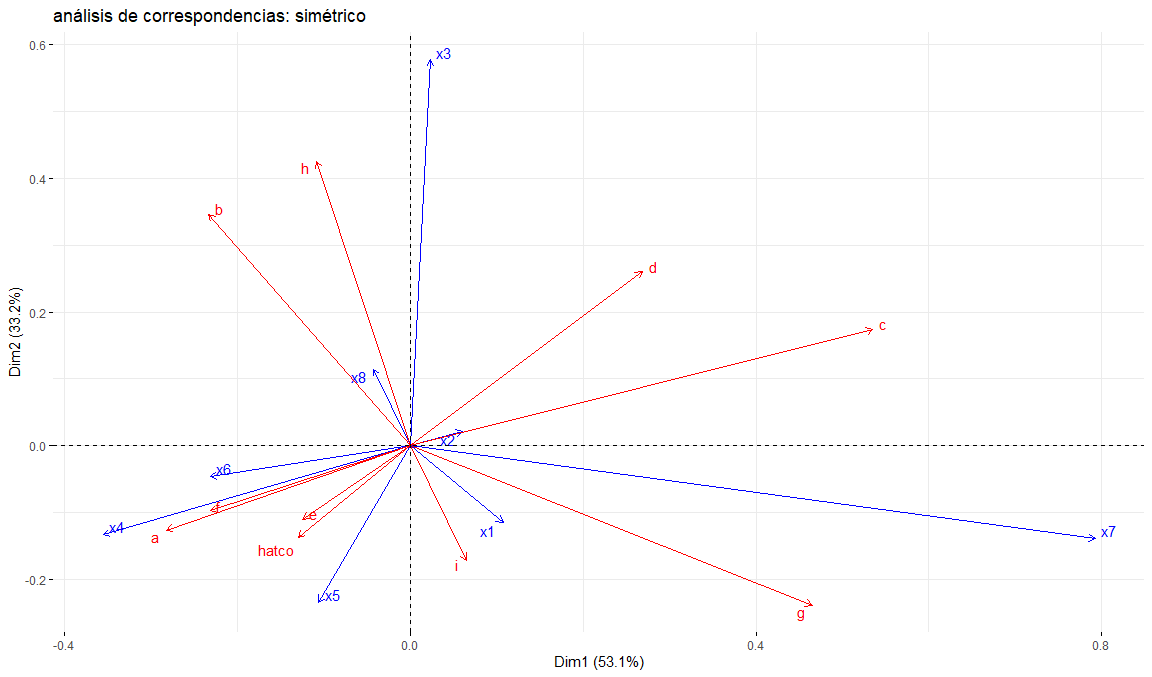
5.5.3 Mapas de puntos
Calculamos el mapa de filas y columnas en el mismo plano y los mapas conjuntos (mapa clásico y relacionados)
fviz_ca_biplot(
res.ca,
map = "symmetric",
title = "análisis de correspondencias: simétrico",
arrow = c(TRUE, TRUE),
repel = TRUE
)
fviz_ca_biplot(
res.ca,
map = "rowprincipal",
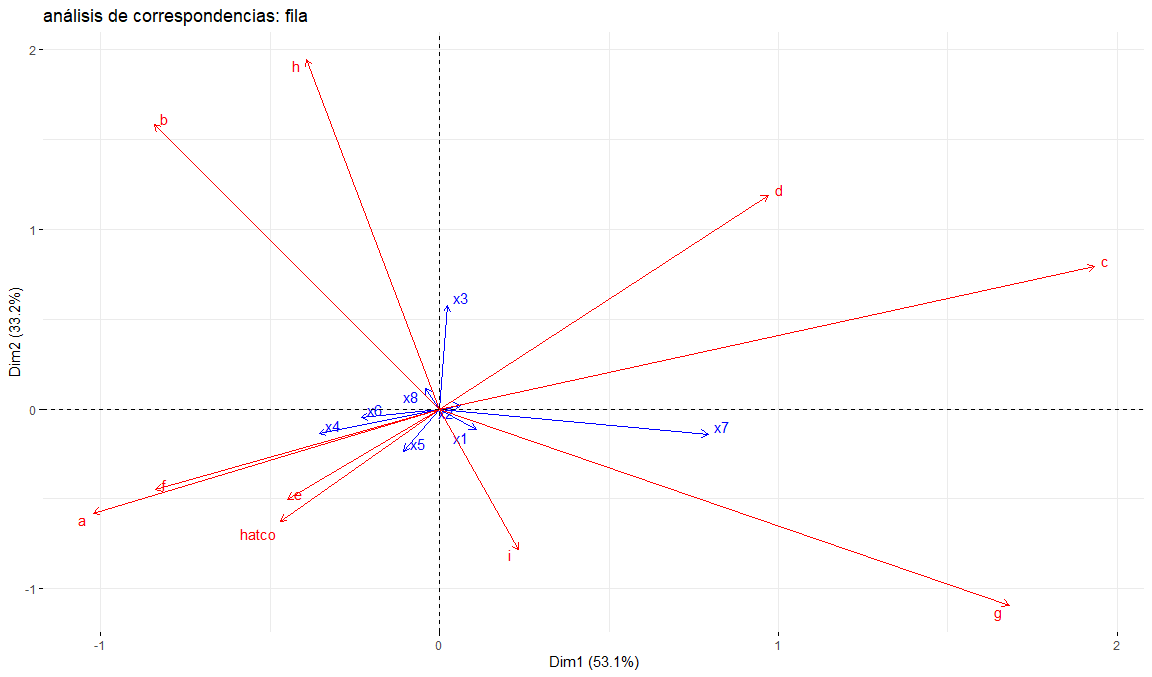
title = "análisis de correspondencias: fila",
arrow = c(TRUE, TRUE),
repel = TRUE
)
fviz_ca_biplot(
res.ca,
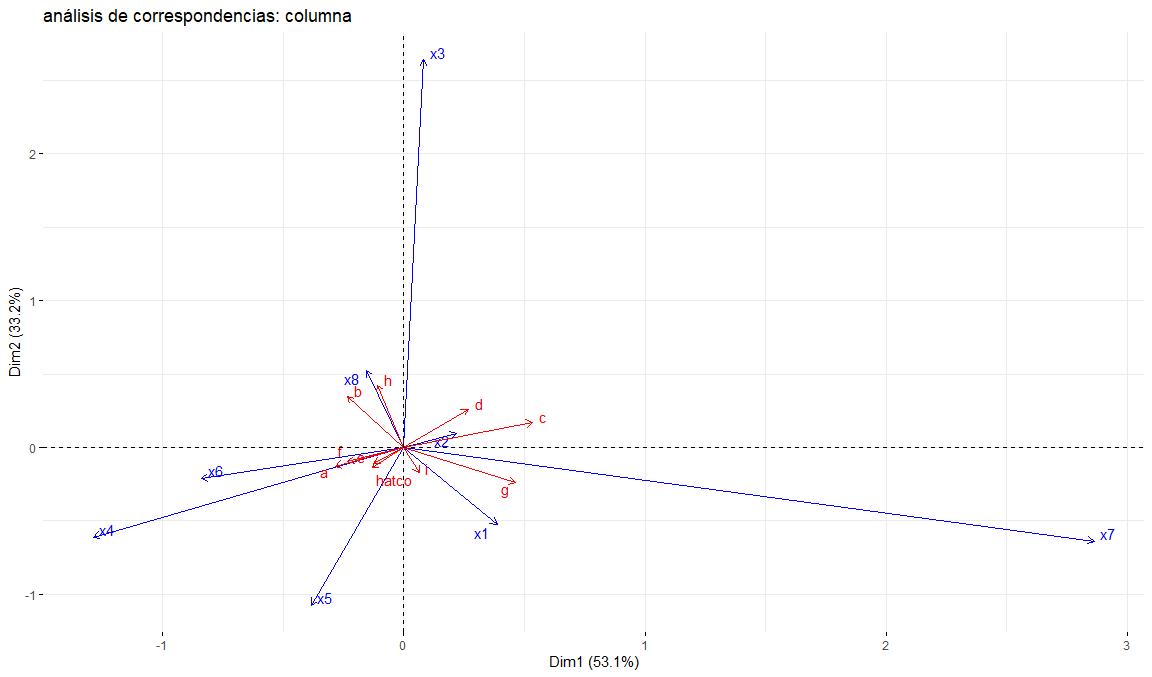
map = "colprincipal",
title = "análisis de correspondencias: columna",
arrow = c(TRUE, TRUE),
repel = TRUE
)
fviz_ca_biplot(
res.ca,
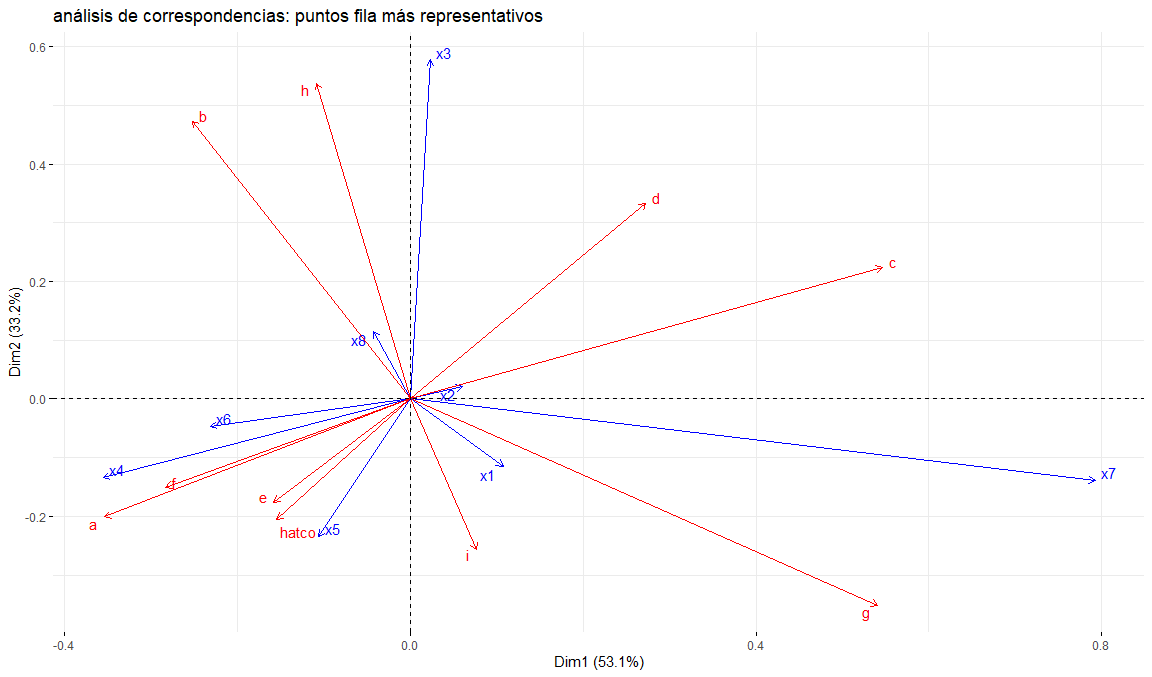
map = "rowgreen",
title = "análisis de correspondencias: puntos fila más representativos",
arrow = c(TRUE, TRUE),
repel = TRUE
)
fviz_ca_biplot(
res.ca,
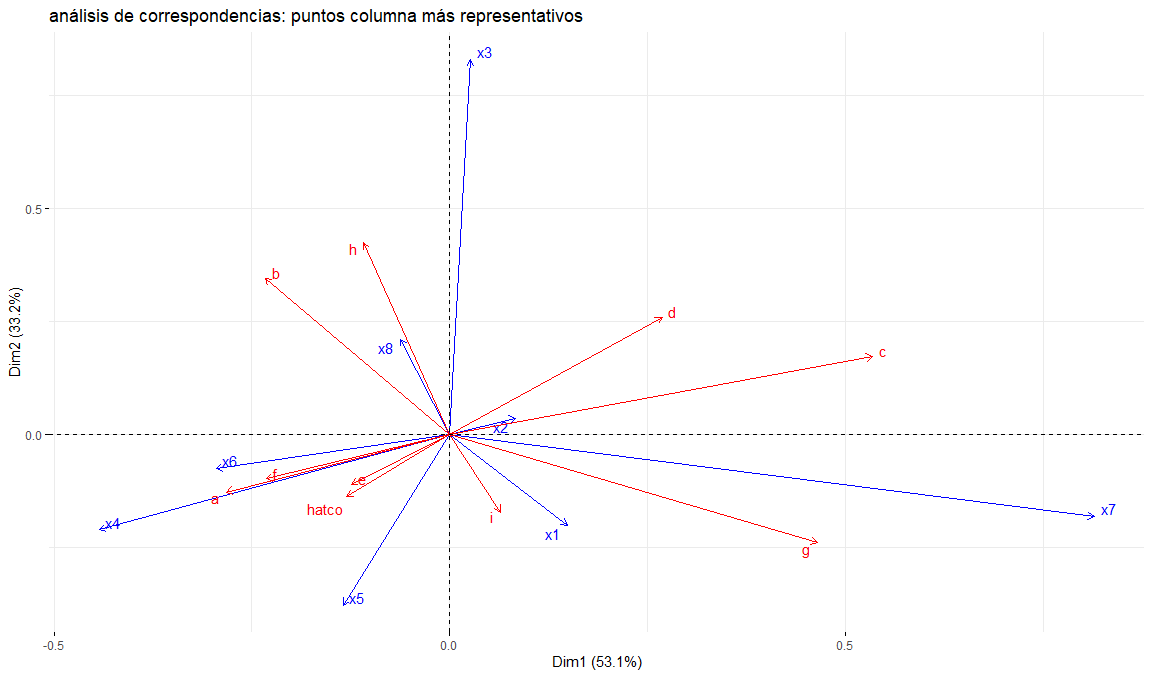
map = "colgreen",
title = "análisis de correspondencias: puntos columna más representativos",
arrow = c(TRUE, TRUE),
repel = TRUE
)
fviz_ca_biplot(
res.ca,
map = "symbiplot",
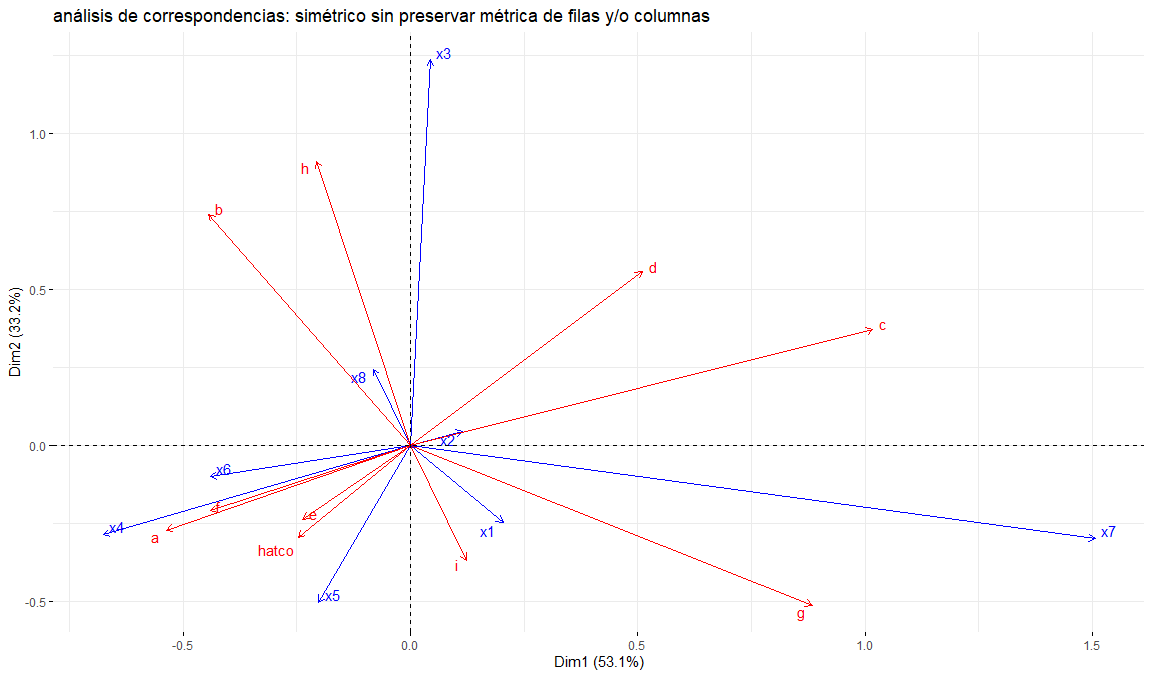
title = "análisis de correspondencias: simétrico sin preservar métrica de filas y/o columnas",
arrow = c(TRUE, TRUE),
repel = TRUE
)
fviz_ca_biplot(
res.ca,
map = "rowgab",
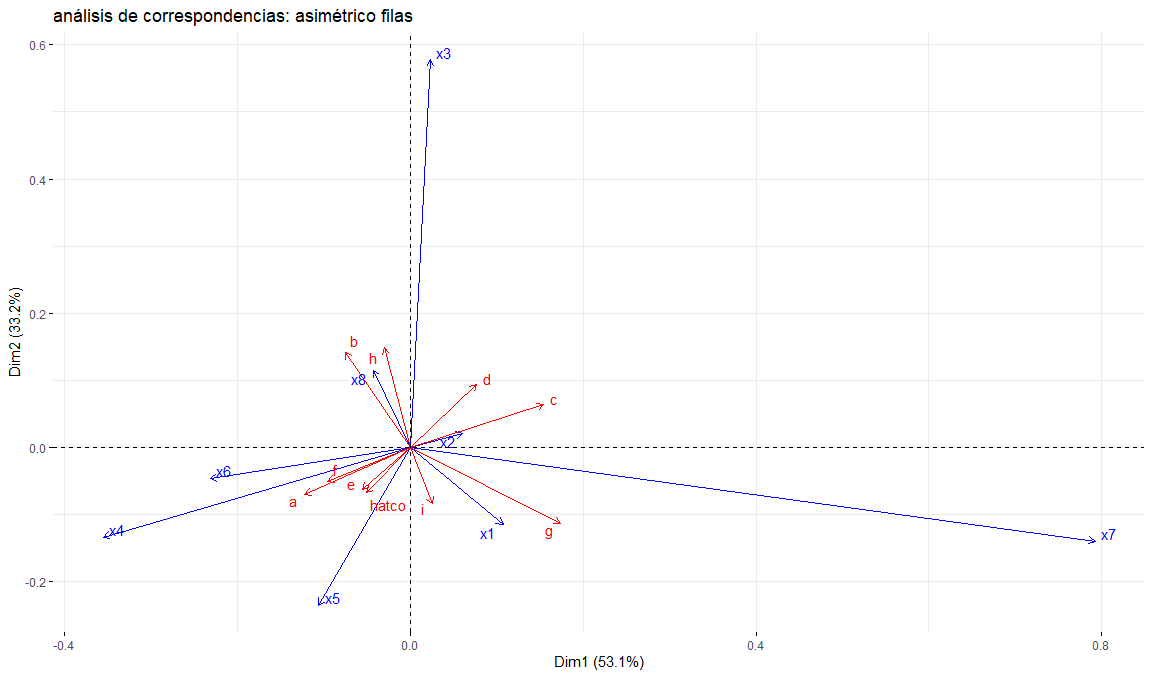
title = "análisis de correspondencias: asimétrico filas",
arrow = c(TRUE, TRUE),
repel = TRUE
)
fviz_ca_biplot(
res.ca,
map = "colgab",
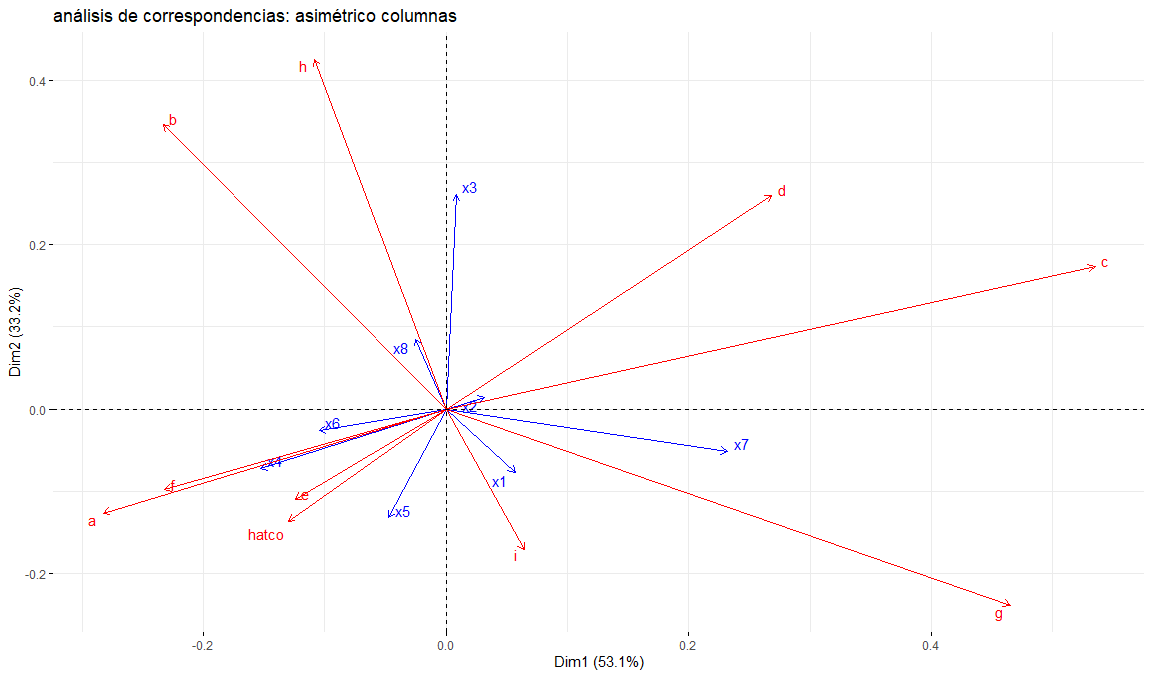
title = "análisis de correspondencias: asimétrico columnas",
arrow = c(TRUE, TRUE),
repel = TRUE
)
5.5.4 Otros gráficos
Calculamos otros gráficos de soporte a la comprensión del análisis
Mapa de filas
fviz_ca_row(res.ca, repel = TRUE, col.row = "blue")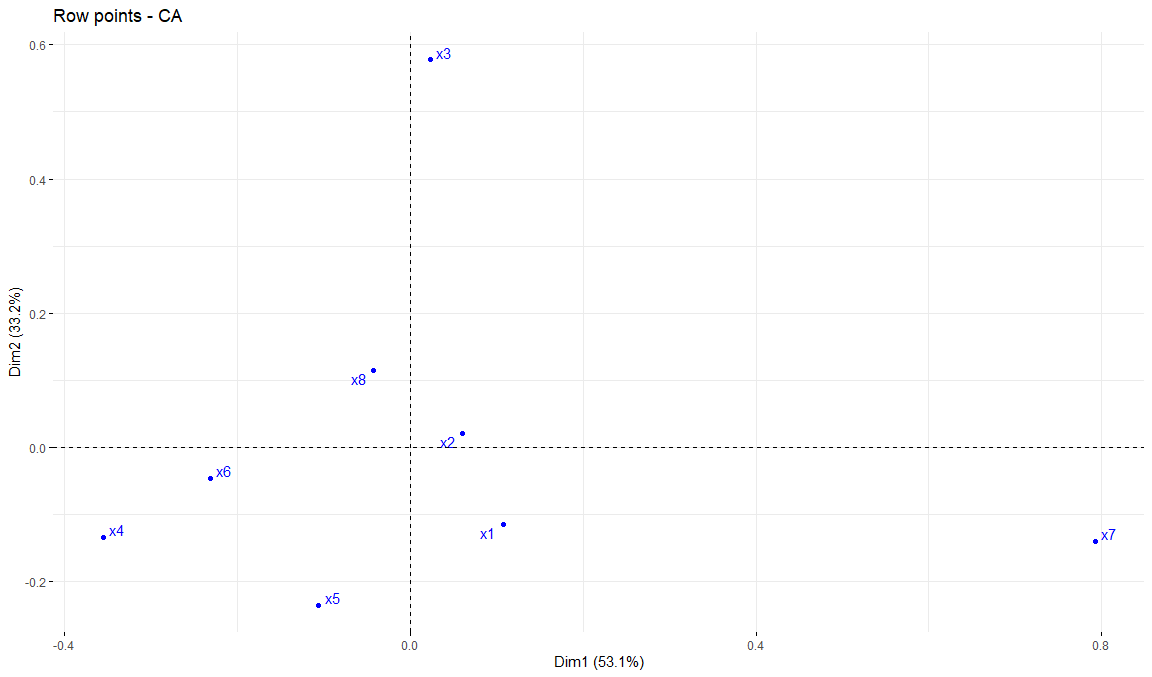
Mapa de columnas
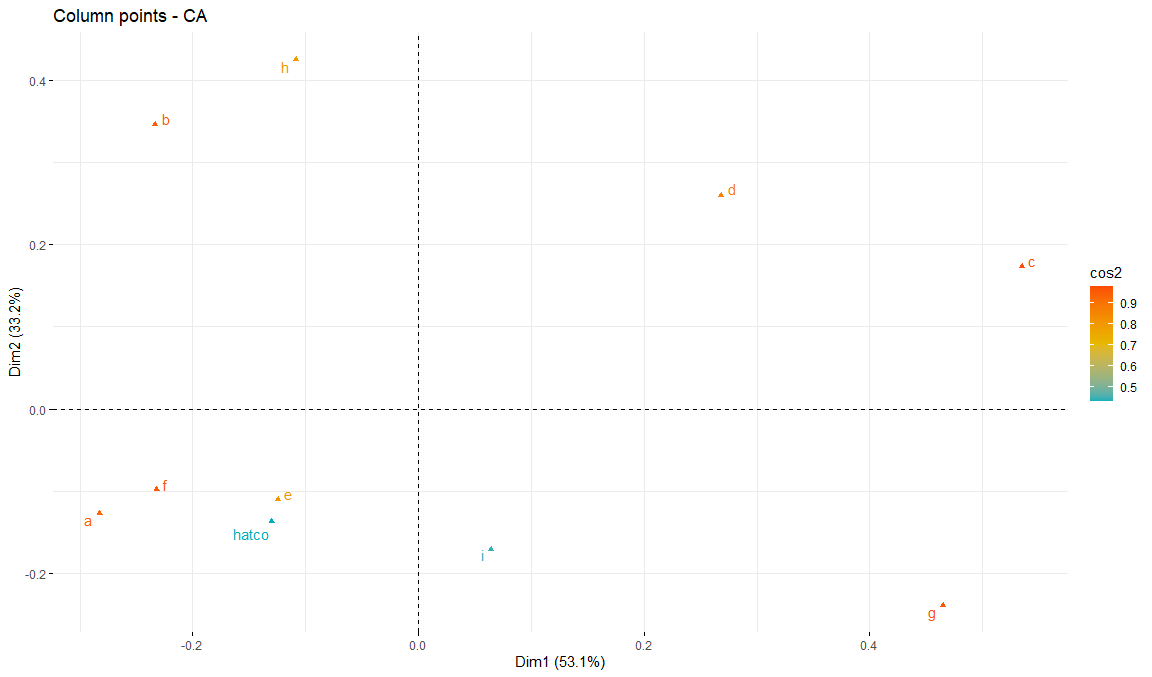
fviz_ca_col(res.ca, repel = TRUE, col.col = "red")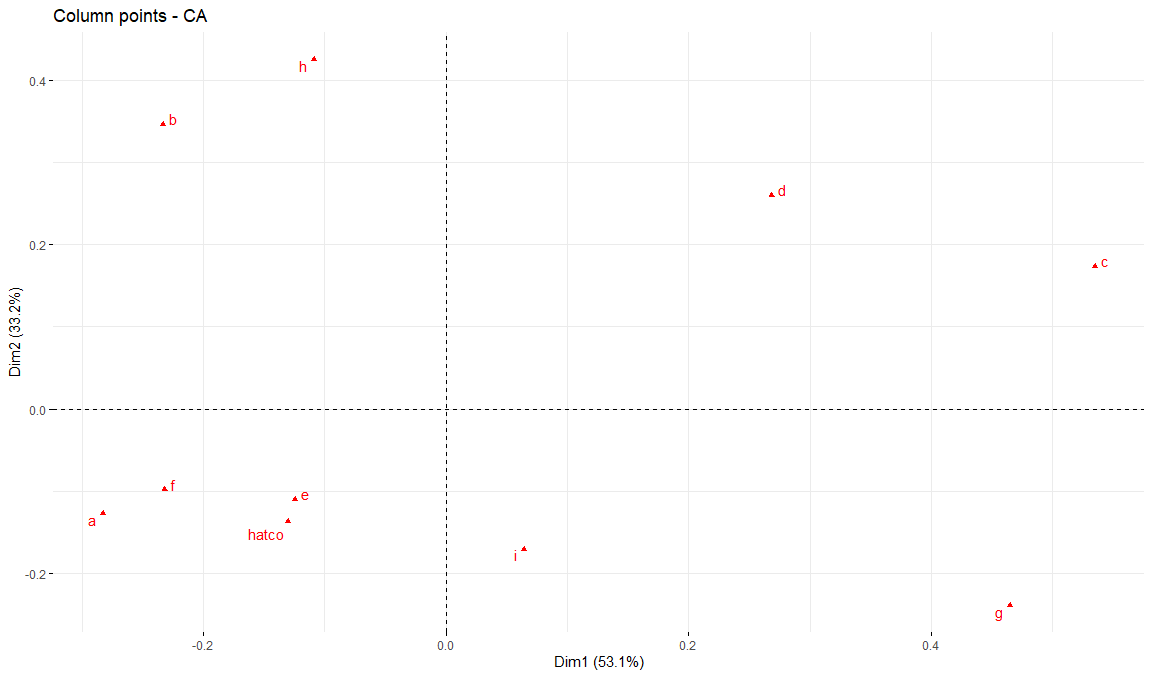
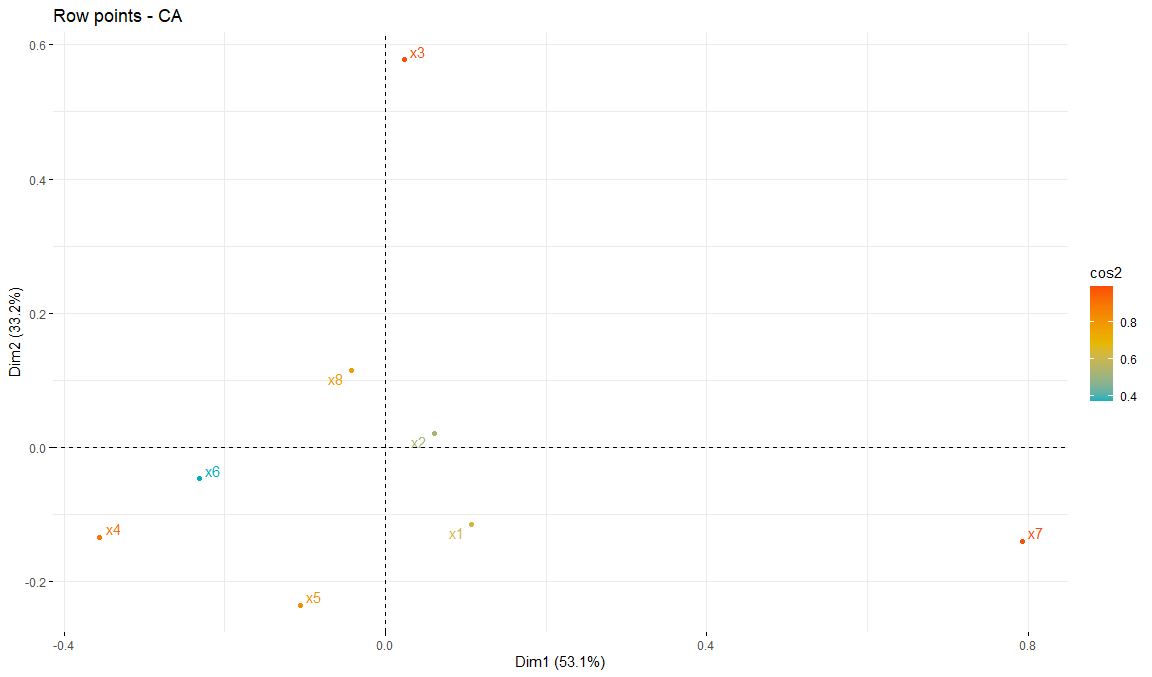
Calidad de puntos columna
fviz_ca_row(
res.ca,
col.row = "cos2",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE
)
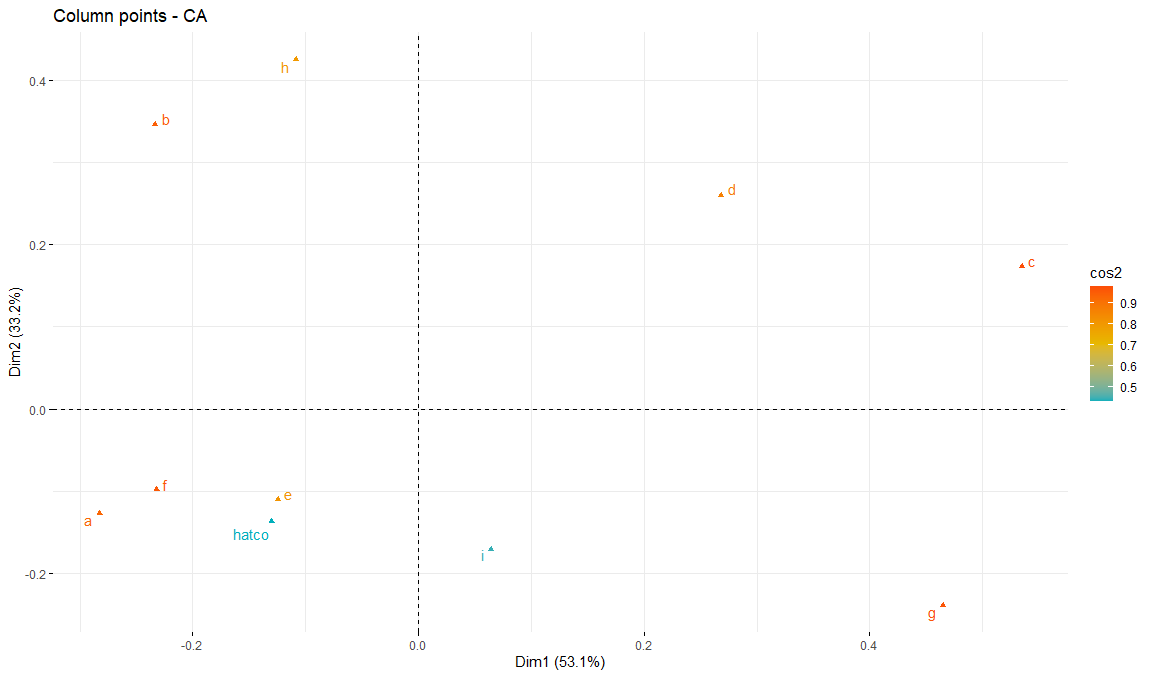
Calidad de puntos fila
fviz_ca_col(
res.ca,
col.col = "cos2",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE
)
Creamos un mapa de correlación entre calidad filas/columnas y dimensiones
corrplot(res.ca[["row"]][["cos2"]], is.corr = FALSE)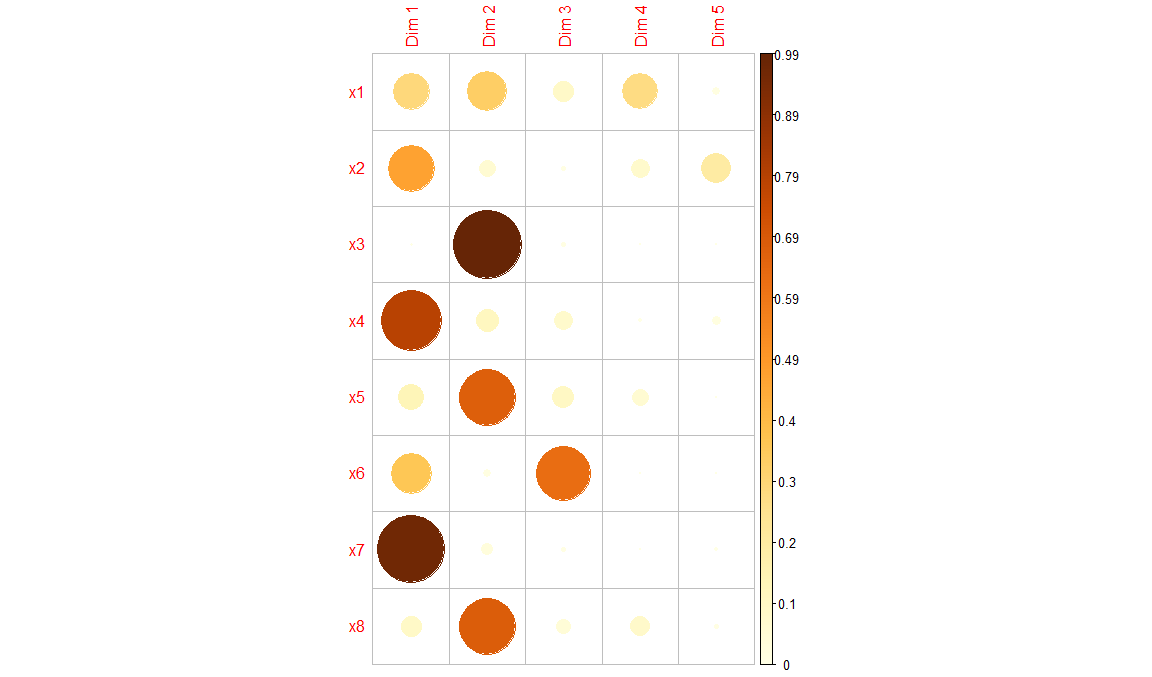
Calidad de representación
corrplot(res.ca[["col"]][["cos2"]], is.corr = FALSE)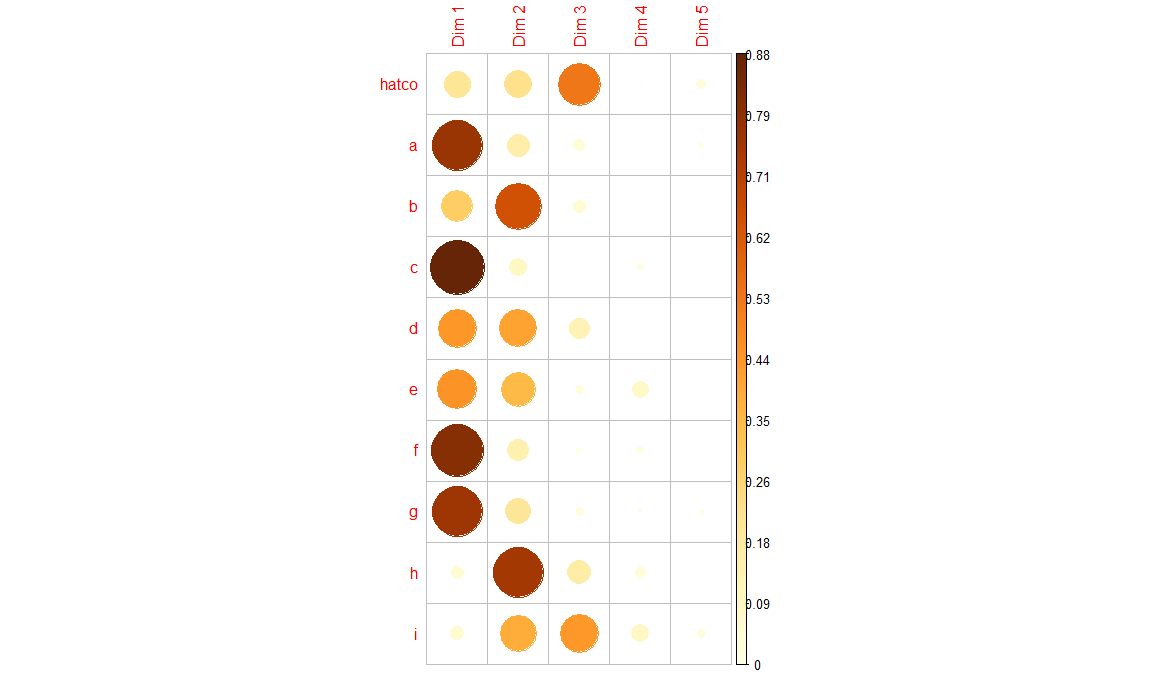
Calidad de filas
fviz_cos2(res.ca, choice = "row", axes = 1:2)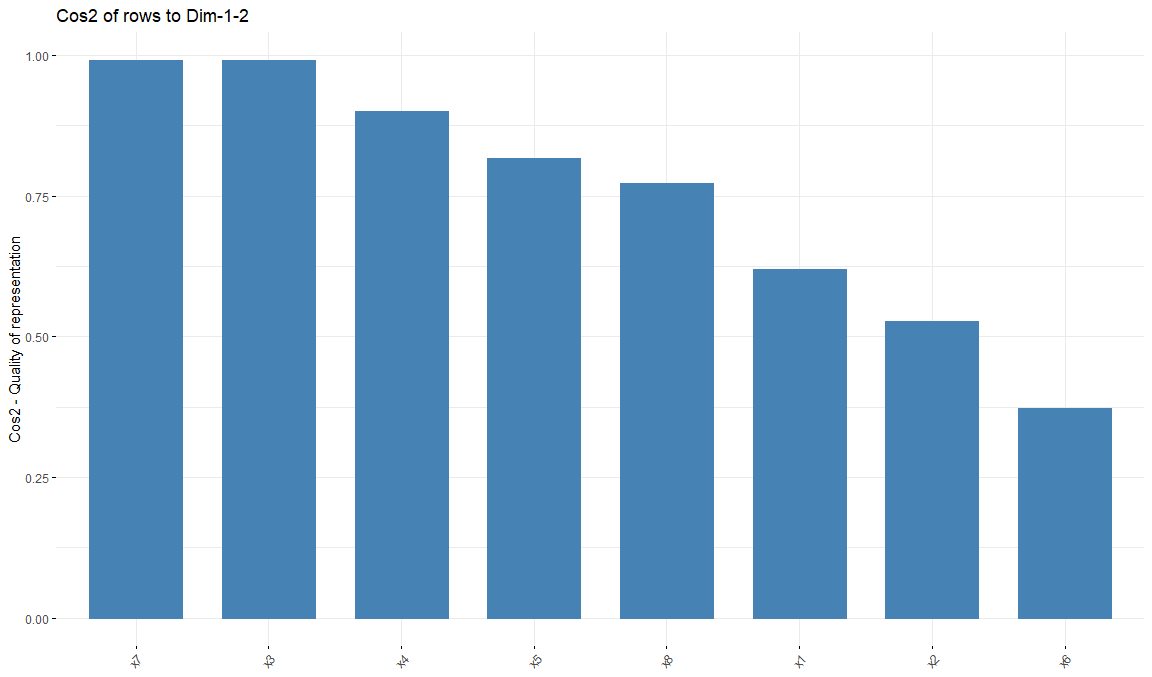
Calidad de columnas
fviz_cos2(res.ca, choice = "col", axes = 1:2)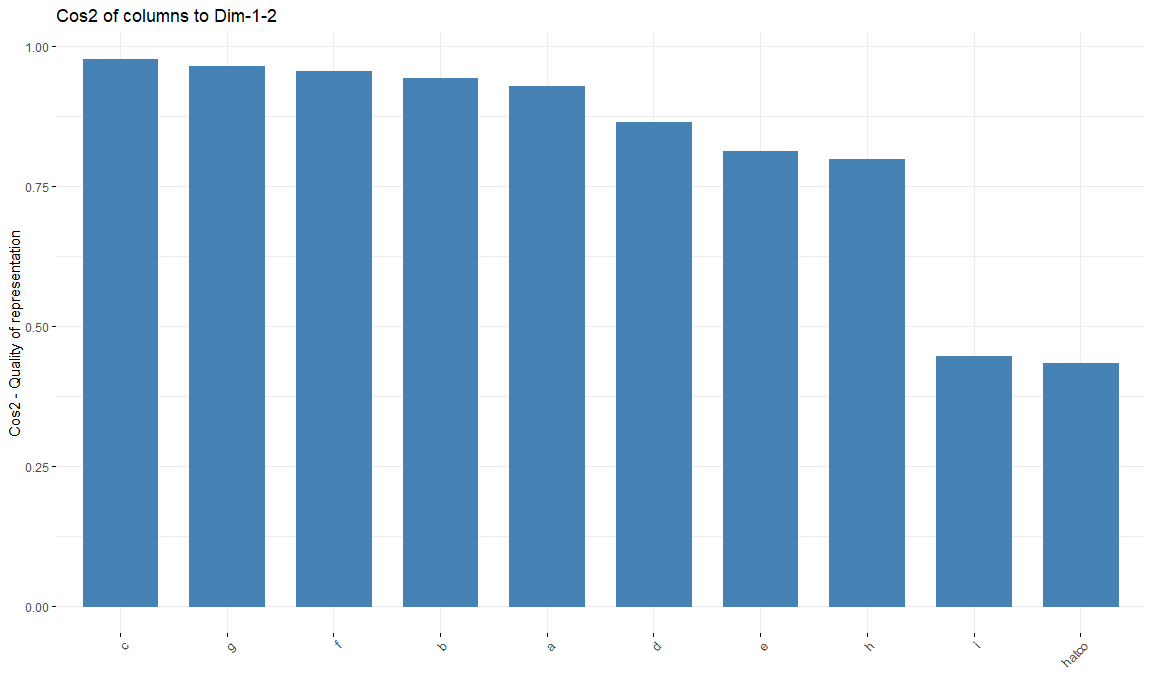
Correlación filas en dimensiones
corrplot(res.ca[["row"]][["contrib"]], is.corr = FALSE)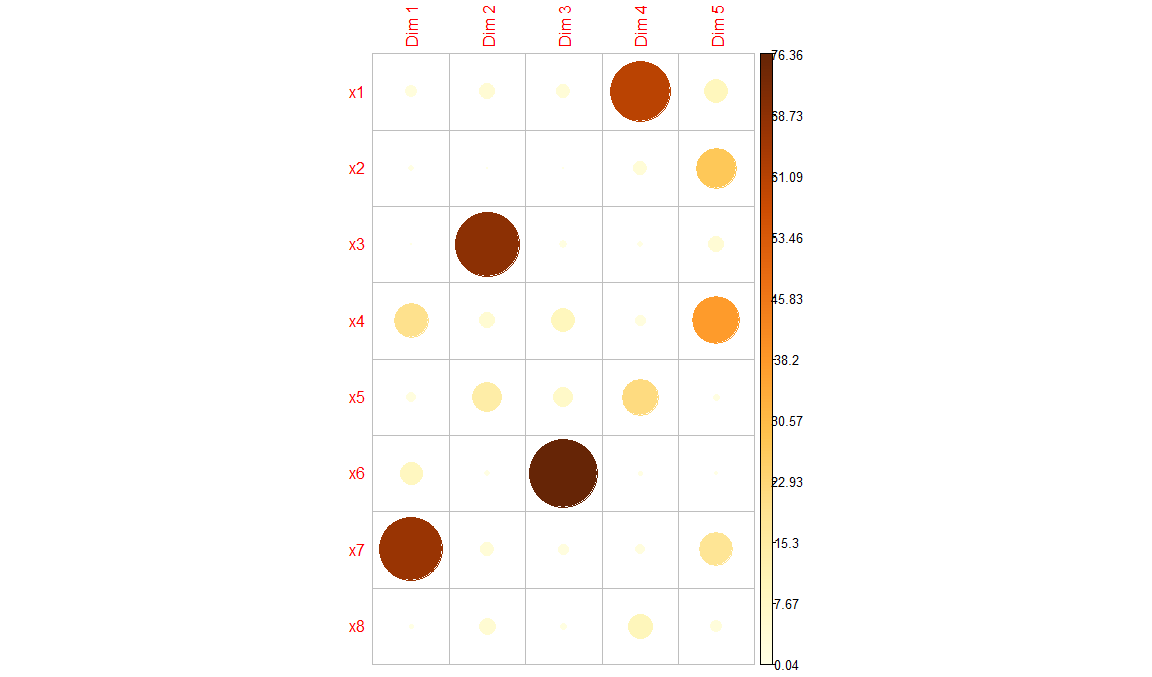
Correlación columnas en dimensiones
corrplot(res.ca[["col"]][["contrib"]], is.corr = FALSE)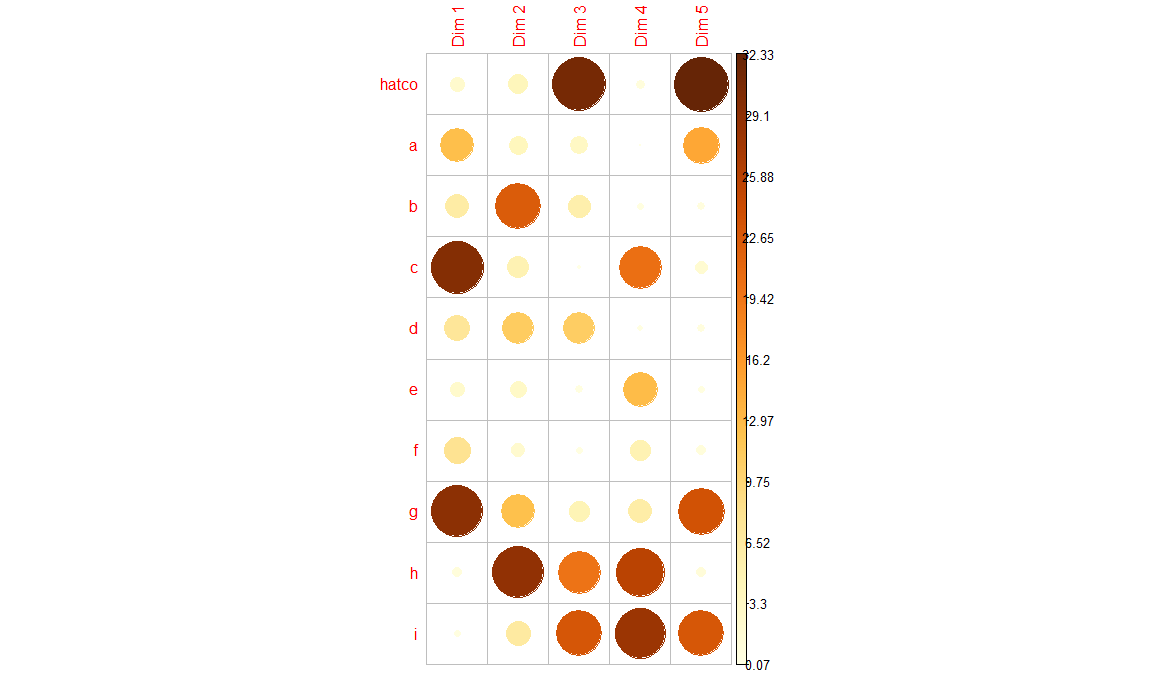
Contribuciones de las filas a la primera dimensión
fviz_contrib(res.ca,
choice = "row",
axes = 1,
top = 10)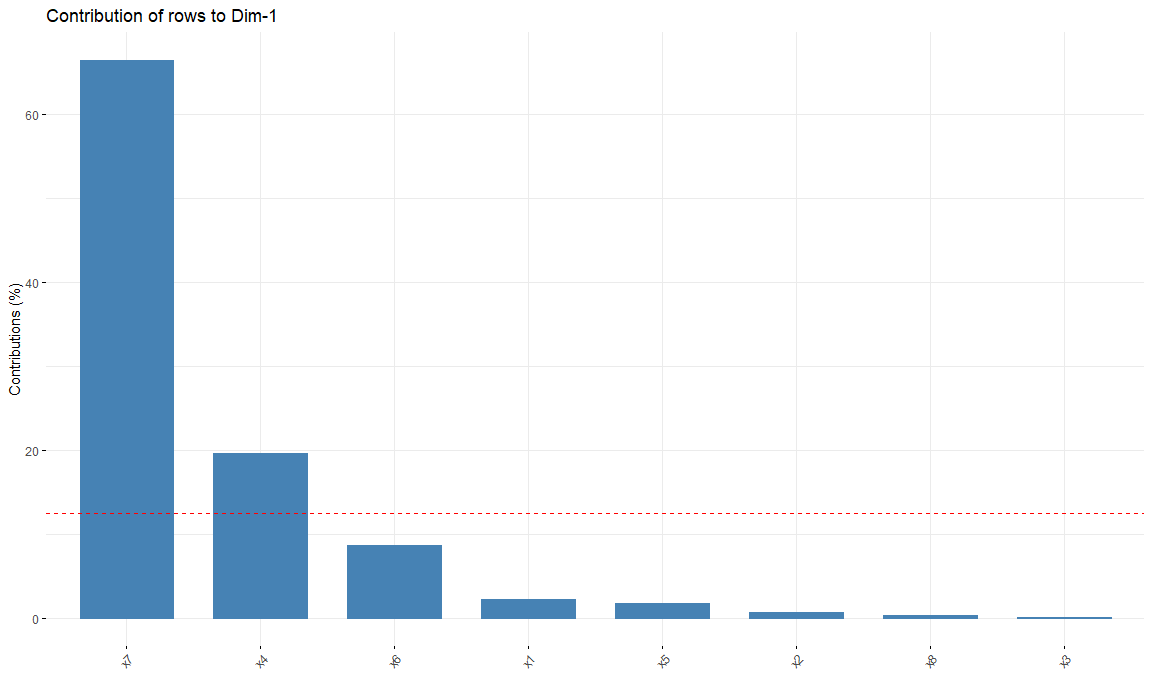
Contribuciones de las filas a la segunda dimensión
fviz_contrib(res.ca,
choice = "row",
axes = 2,
top = 10)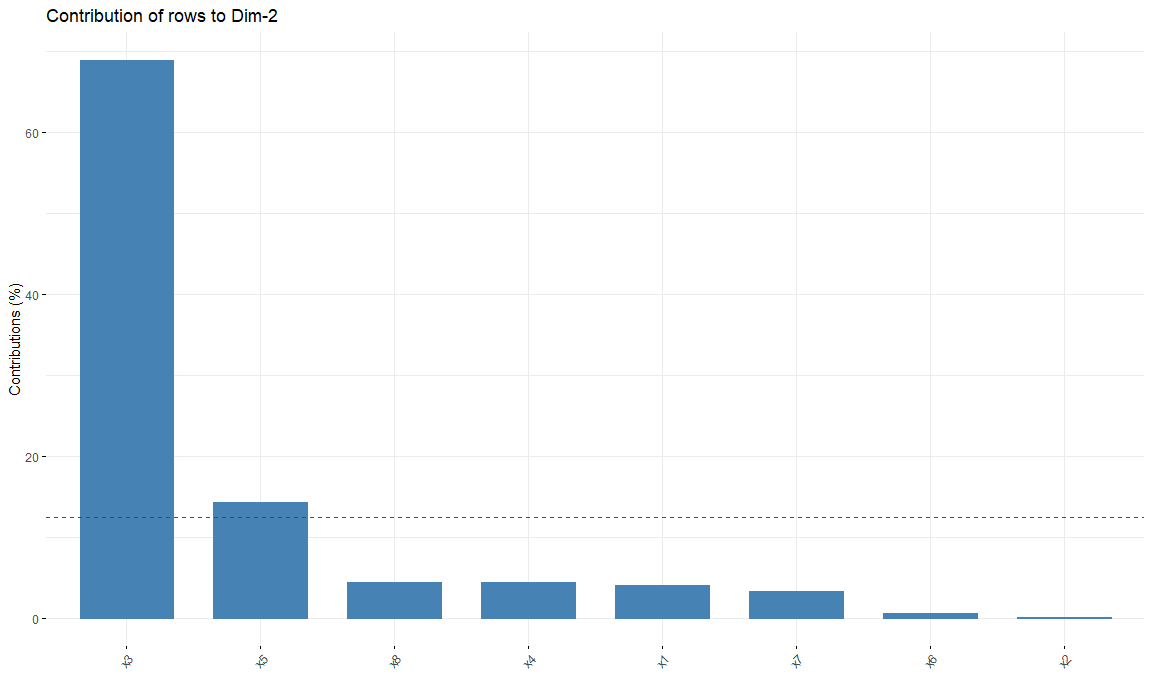
Contribuciones de las columnas a la primera dimensión
fviz_contrib(res.ca,
choice = "col",
axes = 1,
top = 10)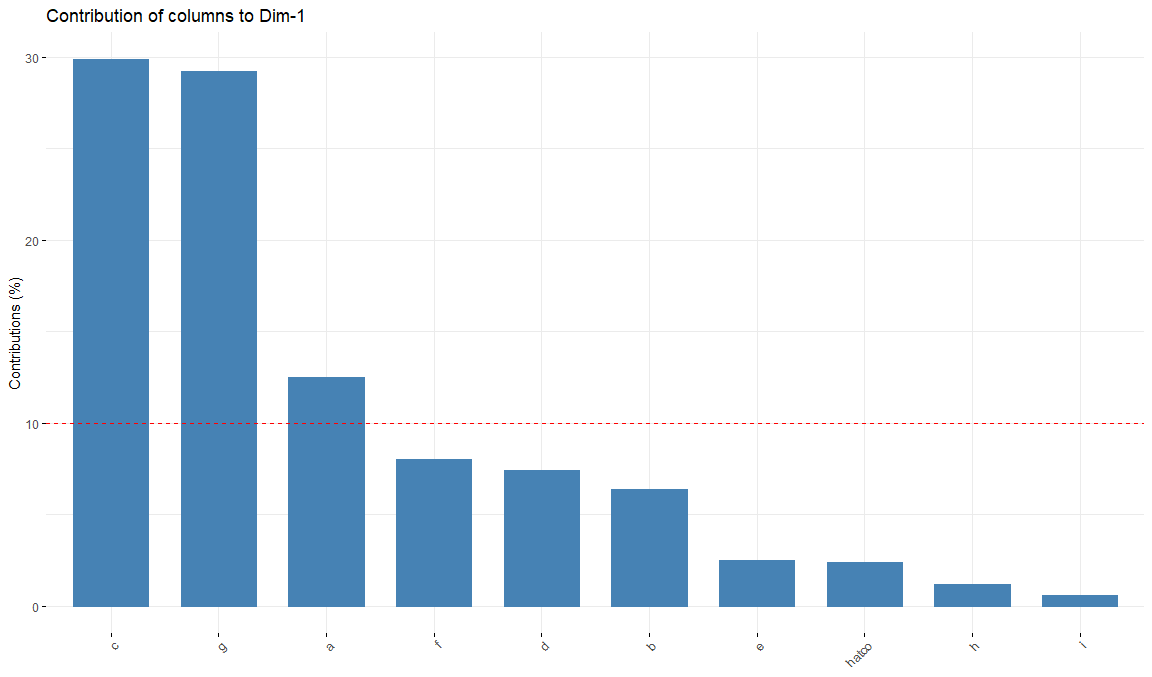
Contribuciones de las columnas a la segunda dimensión
fviz_contrib(res.ca,
choice = "col",
axes = 2,
top = 10)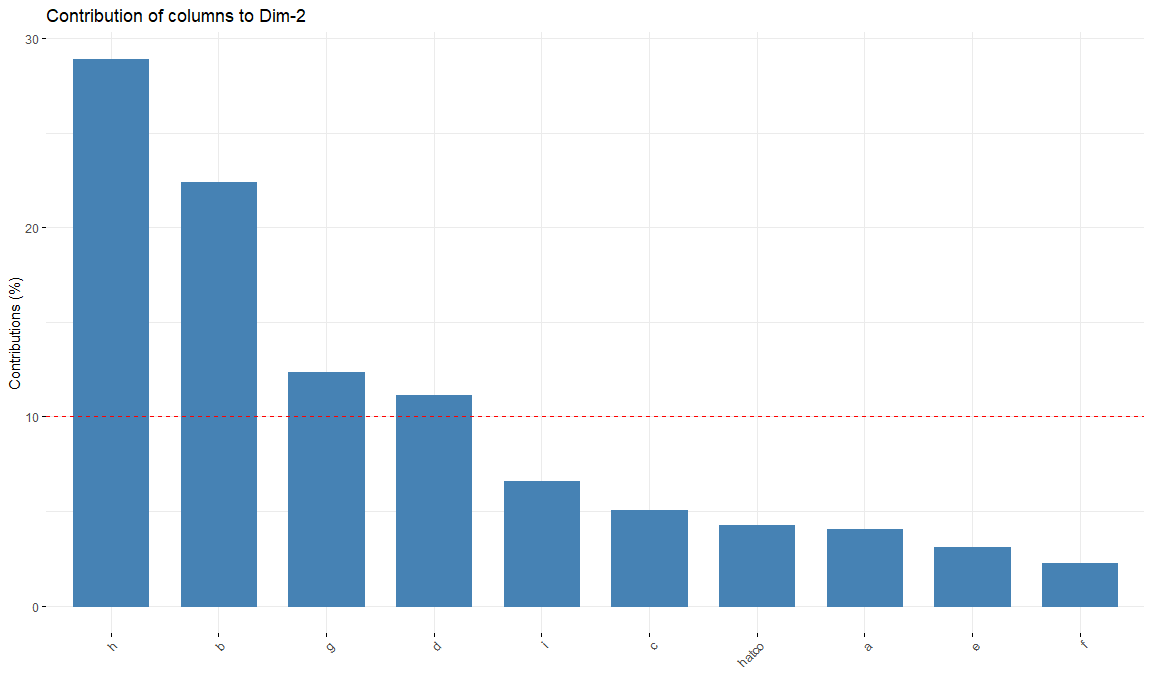
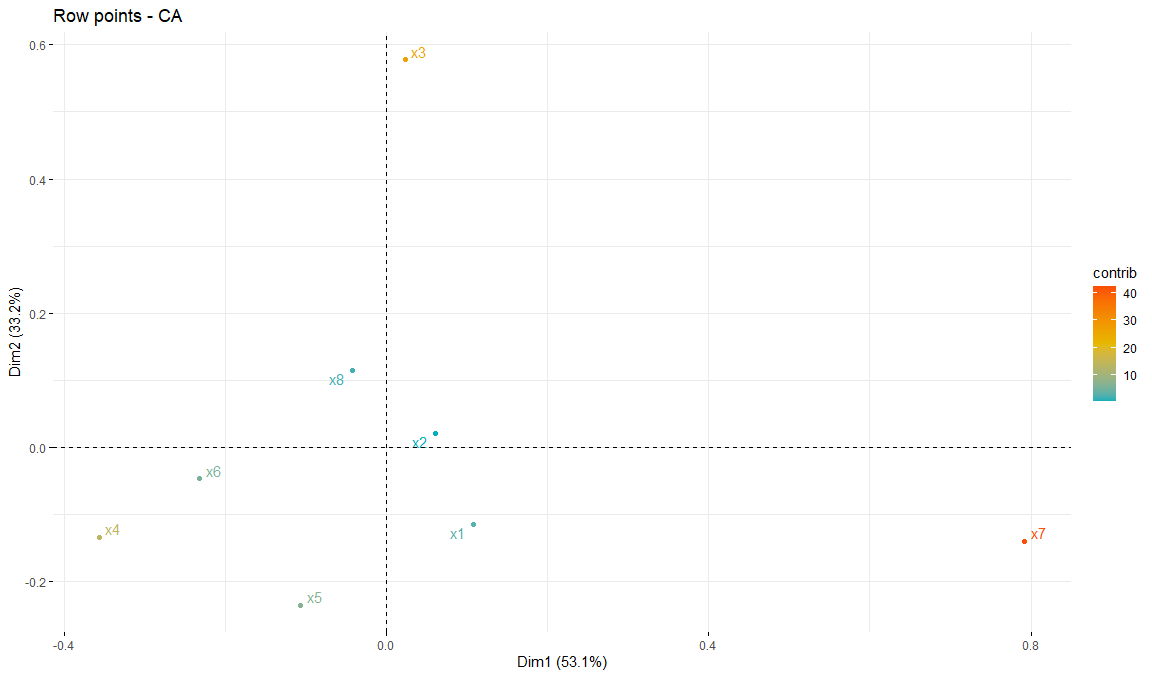
Contribuciones de fila con gradiente
fviz_ca_row(
res.ca,
col.row = "contrib",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE
)
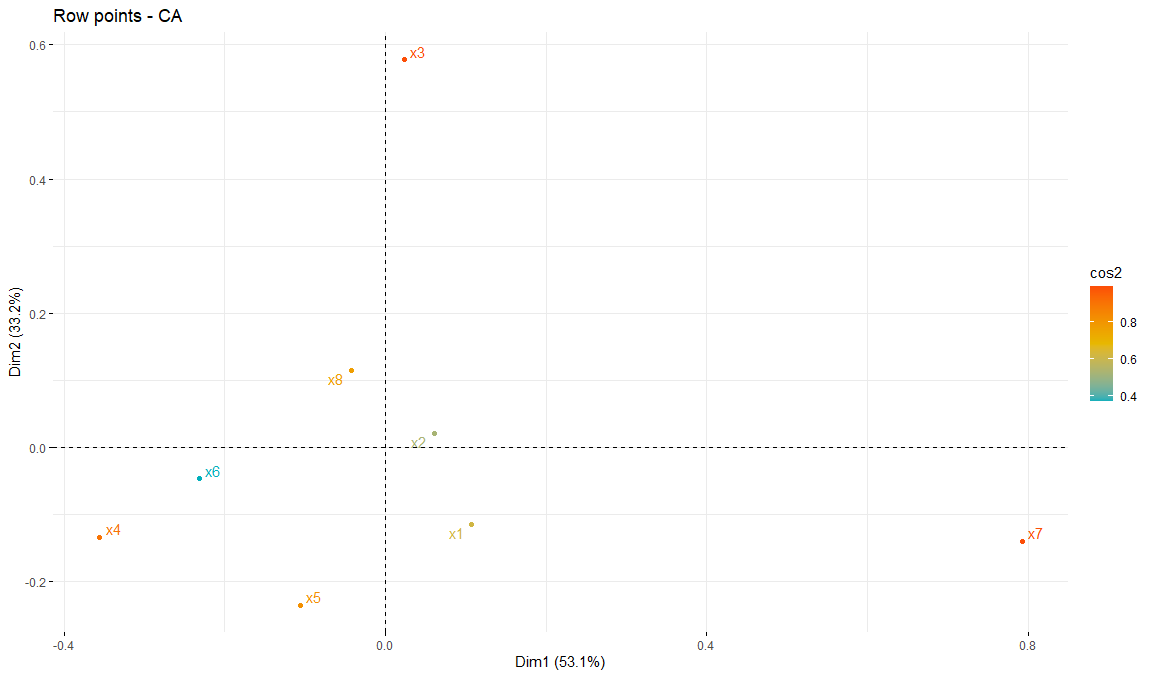
Calidad de fila con gradiente
fviz_ca_row(
res.ca,
col.row = "cos2",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE
)
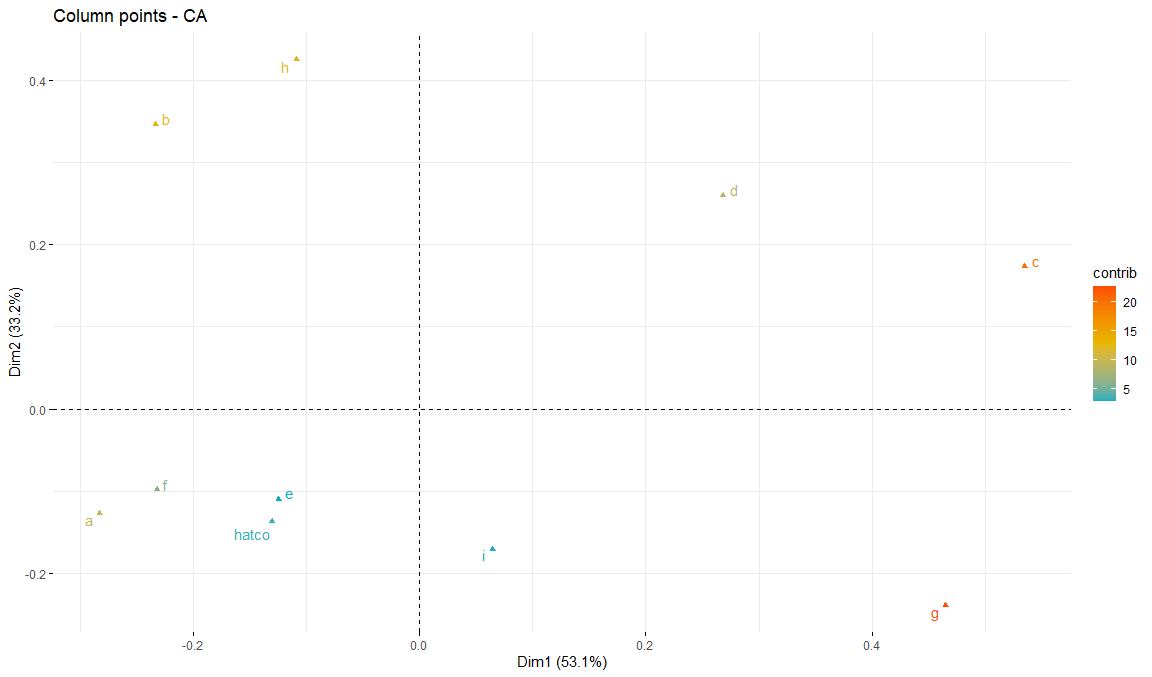
Contribuciones de columna con gradiente
fviz_ca_col(
res.ca,
col.col = "contrib",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE
)
Calidad de columna con gradiente
fviz_ca_col(
res.ca,
col.col = "cos2",
gradient.cols = c("#00AFBB", "#E7B800", "#FC4E07"),
repel = TRUE
)